| Issue |
BIO Web Conf.
Volume 68, 2023
44th World Congress of Vine and Wine
|
|
|---|---|---|
| Article Number | 02020 | |
| Number of page(s) | 7 | |
| Section | Oenology | |
| DOI | https://doi.org/10.1051/bioconf/20236802020 | |
| Published online | 06 December 2023 | |
Do Totiviruses of the veil of Flor yeasts provide an evolutionary advantage?
1 Laboratorio de Microbiología, Departamento de Biomedicina, Biotecnología y Salud Pública, 11510, Puerto Real, Cádiz, España
2 Bodegas Fundador, C. Prta de Rota, S/N, 11408 Jerez de laera, Cádiz
* Corresponding author: gustavo.cordero@uca.es
The main objective of this work was to isolate and characterize viruses that infect both Saccharomyces and other Flor veil-forming genera and suggest the possible consequences they can have on the processes of formation of the Flor veil of typical D.O. Jerez-Xèrés-Sherry wines. Results obtained show that different Flor veil yeast strains isolated in Fino wines and in Amontillados presented a great resistance to ethanol and all of them presented infection by Totiviruses. These were identified after the extraction of double-stranded RNA and amplified by multiplex PCR with specific primers for the most common viruses involved in the killer factor. Subsequently, the same strains of both the S. cerevisiae species and Wickerhamomyces anomalus were subjected to an antiviral treatment with ribavirin. After microbiological analysis with Methylene Blue (MB) plates and by rt-PCR, it was determined that most of them did not show killer factor and the viruses involved in it had been correctly eliminated under the treatment with ribavirin. Subsequently, competition and implantation experiments of the treated and untreated yeasts were carried out against their controls. The wines inoculated with yeasts infected by Totiviruses showed to be dominant in all cases.
© The Authors, published by EDP Sciences, 2023
 This is an Open Access article distributed under the terms of the Creative Commons Attribution License 4.0, which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited.
This is an Open Access article distributed under the terms of the Creative Commons Attribution License 4.0, which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited.
1 Totivirus en levaduras de velo de Flor
Las levaduras son hongos unicelulares que desempeñan un papel crucial en diversas industrias, como la panadería, la cervecería y la enología. Estos microorganismos también se han convertido en importantes organismos modelo para estudiar procesos biológicos fundamentales. Sin embargo, investigaciones recientes han arrojado luz sobre un aspecto menos conocido de la biología de las levaduras: su interacción con los Totivirus [1–2]. Las levaduras de velo de Flor están adaptadas a ambientes hostiles, se forma en vinos con bajo pH y con altas concentraciones de etanol (> 15.5% v/v).
Los Totivirus son pequeños virus de ARN bicatenario que infectan a amplio espectro de especies de hongos, incluidas las levaduras. Este trabajo tiene como objetivo explorar la fascinante relación entre las levaduras y los Totivirus, destacando, por primera vez, su impacto en la biología de las levaduras de velo de Flor y sus posibles aplicaciones en bodegas del Marco de Jerez.
1.1 Interacción entre las levaduras y los Totivirus
Los Totivirus infectan a las levaduras mediante la integración de su genoma de ARN bicatenario en el citoplasma de la célula huésped. Una vez dentro de la célula de levadura, el Totivirus utiliza la maquinaria molecular del huésped para replicar su genoma y producir proteínas virales. Curiosamente, la mayoría de los Totivirus no causan un daño grave a sus hospedadores de levaduras. En cambio, establecen una relación simbiótica, conocida como infección persistente, en la que el virus coexiste con la levadura sin causar un daño significativo [2].
Se ha descubierto que la infección totiviral persistente en las levaduras confiere varias ventajas. Un beneficio notable es el aumento de la tolerancia a diversos estreses ambientales. Algunos estudios han demostrado que las células de levadura infectadas con Totivirus muestran una mayor resistencia a fluctuaciones de temperatura, estrés oxidativo y compuestos tóxicos. Este fenómeno sugiere que la infección totiviral desencadena una respuesta protectora en las levaduras, posiblemente a través de la activación de genes relacionados con el estrés. Comprender los mecanismos moleculares detrás de este efecto protector podría tener implicaciones en aplicaciones biotecnológicas, como el desarrollo de cepas de levaduras tolerantes al estrés para procesos industriales [3].
1.2 Impacto en la biología de las levaduras
La infección totiviral también puede influir en aspectos fundamentales de la biología de las levaduras. Por ejemplo, se ha observado que la presencia de Totivirus puede alterar la velocidad de crecimiento y la morfología de las levaduras. Las células infectadas pueden mostrar cambios en el tamaño celular, la forma y los patrones de crecimiento. Además, los Totivirus pueden afectar la expresión de ciertos genes en las levaduras, lo que conlleva alteraciones en las vías metabólicas y las funciones celulares. Estos hallazgos proporcionan valiosos conocimientos sobre la interacción intrincada entre la infección viral y la biología del hospedador, ampliando nuestra comprensión de la genética y fisiología de las levaduras [4].
La relación única entre las levaduras y los Totivirus ha abierto nuevas vías en la investigación biotecnológica. La interacción entre las levaduras y los Totivirus representa un área de investigación fascinante en el campo de la biología de las levaduras. La relación simbiótica entre estos microorganismos resalta la complejidad de las interacciones huésped-virus y su impacto en los procesos celulares. Investigaciones adicionales sobre los mecanismos subyacentes de la infección totiviral en las levaduras pueden revelar nuevas estrategias para mejorar las aplicaciones basadas en levaduras en diversas industrias. A medida que nuestro entendimiento se profundiza, el potencial para aprovechar el poder de las levaduras y los Totivirus en biotecnología sin duda se expandirá, dando lugar a avances emocionantes en el campo [4–6].
El objetivo principal de este trabajo fue aislar y caracterizar virus que infectan tanto a Saccharomyces como a otros géneros que forman el velo de Flor, y sugerir las posibles consecuencias que pueden tener en los procesos de formación del velo de Flor en los vinos elaborados bajo crianza biológica de la D.O. Jerez-Xèrés-Sherry.
2 Materiales y métodos
2.1 Plan de muestreo y aislamiento de levaduras de velo de Flor
2.1.1 Plan de muestreo
Las muestras se tomaron de diversas botas de tres bodegas ubicadas en Jerez de la Frontera, una en El Puerto de Santa María y otra en Sanlúcar de Barrameda, con crianza biológica en sistema de criaderas y soleras y una en sistema de crianza oxidativa (Amontillado) en estudios anteriores [7–9]. En este estudio, el muestreo se realizó en Bodegas Fundador (Jerez de la Frontera, España) en 9 botas de vinos Finos diferentes. Para ello se utilizó una venencia modificada estéril a modo de red para recoger el velo sin dañarlo. Entre muestra y muestra se lavaba la venencia con etanol al 70% y suero fisiológico para evitar contaminaciones cruzadas.
Cada muestra, se nombró por el número de andana, número de bota y tipo de crianza. Las muestras se depositaron frascos de plástico estériles, los cuales fueron transportados en nevera portátil al laboratorio para evitar alteraciones en las levaduras por las condiciones externas.
2.1.2 Aislamiento de levaduras de velo de Flor
En el laboratorio, las muestras fueron introducidas en tubos Falcon estériles donde se centrifugaron a 3000 rpm por 5 minutos descartando el sobrenadante, obteniendo un pellet de levaduras. Los pellets fueron resuspendidos en 10 ml de agua estéril para volver a centrifugar durante 5 minutos a 3000 rpm para terminar de limpiar la muestra. El sobrenadante fue descartado y se resuspendió en 1 ml de agua estéril en tubos Eppendorf de 1,5 ml.
Las muestras lavadas fueron sembradas en estría, bajo condiciones de esterilidad en placas de Petri con medio de cultivo comercial WL (Condalab, Madrid, España). Se incubaron en posición invertida a una temperatura de 28ºC durante 24-48 horas.
Posteriormente, se seleccionaron colonias para ser aisladas como colonias puras en placas de Petri con medio de cultivo YPD (1% extracto de levadura, 2% peptona, 2% glucosa, 2% agar), bajo condiciones estériles. Las placas de Petri fueron incubadas en posición invertida a una temperatura de 28 ºC durante 24-48 horas. Posteriormente, se seleccionaron 30 colonias al azar conforme a su morfología y dimensión, y se glicerinaron para conservarlas a -20 ºC.
2.2 Extracción de ADN de las levaduras, identificación por PCR-ITS y SSR-PCR
La identificación de las colonias se llevó a cabo mediante la obtención de los diferentes perfiles moleculares del ADN total previamente extraído mediante protocolos estándares de extracción. Posteriormente, se cuantificó en ADN en un Nanodrop y se identificaron por las técnicas de biología molecular PCR de la región ITS del ADN ribosómico [10] y RFLP utilizando las endonucleasas de restricción HaeIII, HinfI y CfoI [11]. Aquellas levaduras identificadas como S. cerevisiae, se identificaron a nivel de cepa empleando la técnica del análisis de los microsatélites multiplex o SSR [12,13]. Todos los productos amplificados se sometieron a electroforesis en geles de agarosa o en secuenciador automático. El análisis de las imágenes se llevó a cabo mediante la utilización de una cámara equipada con un transiluminador UV (BioRad). Se seleccionaron aquéllas que presentaron un perfil distinto y se tomaron al menos dos representantes de la misma especie para ser secuenciadas. La secuenciación se llevó a cabo a través de los servicios de secuenciación MACROGEN Inc. Europa (http://www.macrogen.com). La alineación de sencuencias se realizó con programas de bioinformática online.
2.3 Determinación del factor killer de las diferentes especies y cepas de levaduras identificadas
Todas las colonias previamente seleccionadas e identificadas fueron sometidas a evaluación de su factor killer. Para ello se preparó un medio de cultivo específico con azul de metileno, YPD-MB (1% extracto de levadura, 2% peptona, 2% glucosa, 3% NaCl, 2% agar) en un tampón citrofosfato 0,1 M. El pH de este medio se ajustó a 4,7. Tras esterilizar el medio en autoclave a 121 ºC durante 20 minutos, se dejó bajar la temperatura a 55 ºC y se le añadió una solución de azul de metileno al 1.5% (v/v), aproximadamente 4 mg/ml.
Una vez preparadas las placas de YPD-MB, se sembró en cada una de ellas en césped una cepa de S. cerevisiae sensible K0. Posteriormente se procedió a sembrar en gota (spotting) 10 µL de cada cepa (previamente cultivadas en YPD líquido durante 24-48 horas), a razón de 8 cepas por placa, para comprobar su actividad killer. En el centro de cada placa, a modo de control, se añadió una gota de 10 µL de una cepa de actividad killer positivo conocido, en concreto la cepa K1.
Tras esto, se incubó a 28 ºC durante 48h, hasta que se observaron halos de inhibición claros en las cepas que presentaban actividad killer, seleccionándose estas cepas para los siguientes pasos, y descartándose aquellas en las que no se observó actividad killer de forma cualitativa. Además, se midió el diámetro de los halos en mm para obtener de forma semi-cuantitativa el potencial killer de cada cepa que arrojó un resultado positivo. Se seleccionó, además, una cepa de levadura de velo de Flor de la especie S. cerevisiae para utilizarla como control en las pruebas de qPCR posteriores.
2.4 Extracción y cuantificación del ARN viral de las levaduras con fenotipo killer
Las levaduras que mostraron fenotipo killer, una killer negativo como control en las placas de YPD-MB se inocularon en 5 ml de YPD líquido y se incubaron a 28 ºC durante 48 horas, hasta que se obtuvo la cantidad deseada de biomasa.
Para extraer el ARN, se utilizó el kit comercial Rneasy Plus Mini kit (Qiagen, Hilden, Alemania), siguiendo los pasos que indica el protocolo del fabricante. Se prestó máxima atención a las condiciones de esterilidad, y se autoclavaron dos veces todos los materiales a utilizar con el fin de evitar la contaminación con Rnasas.
Tras llevar a cabo la extracción, se procedió a cuantificar el ARN, para lo que se utilizó un fluorímetro, Qubit 4 Fluorometer (Thermo Fisher Scientific, Waltham, EEUU) siguiendo las recomendaciones del fabricante. Posteriormente, se realizó un tratamiento con la enzima DNAsa RQ1 Rnase-free Dnase (Thermo Fisher Scientific, Waltham, EEUU), siguiendo el protocolo indicado por el producto.
2.5 RT-PCR y PCR-Multiplex para la identificación de Totivirus en levaduras de velo de Flor
Como primer paso de la RT-PCR, el ARN extraído a cDNA. Para ello se empleó el Kit comercial PrimeScript RT reagent kit (Perfect Real Time) (Takara Bio Inc, Kasatsu, Japón).
Una vez obtenidos los cDNA de cada una de las cepas objeto de estudio se cuantificó el ADN utilizando el Qubit de acuerdo al procedimiento explicado anteriormente. Seguidamente, se procedió a realizar una PCR multiplexada de acuerdo al protocolo descrito en Quintero-Blanco et al. [14]. A continuación, se muestran los primers utilizados, así como las condiciones del termociclador:
El protocolo de amplificación seguido en el termociclador fue el siguiente: precalentamiento a 94 ºC durante 2 minutos, 30 ciclos a 94 ºC durante 45 segundos, 58 ºC durante 30 segundos, 72 ºC durante 20 segundos, y un último paso a 72 ºC durante 5 minutos.
Relación de primers utilizados para la amplificación del genoma vírico relativo a los Totivirus de levaduras.
2.6 Tratamiento con ribavirina para la eliminación de Totivirus en levaduras de velo de Flor
Con el fin de eliminar los micovirus presentes en las levaduras con actividad killer, se prepararó un medio YPD líquido suplementado con diferentes concentraciones de ribavirina. Estas concentraciones fueron; 100 μM, 200 μM, y 300 μM. Las levaduras seleccionadas fueron tratadas durante 72 horas, posteriormente se procedió de nuevo a realizar una caracterización de su fenotipo killer en medio YPD-MB, así como mediante PCR multiplexada, de la misma manera explicada en los apartados anteriores, con el fin de determinar la curación de las levaduras de los Totivirus presentes en las mismas.
2.7 Ensayos de competencia mediante crianza biológica a escala de laboratorio y seguimiento de la formación de velo de Flor
Posterior a la caracterización molecular de las levaduras y su posterior tratamiento con ribavirina. Se realizaron ensayos de competencia a escala de laboratorio, utilizando la misma cepa de levadura con la presencia de Totivirus y sin ella una vez tratados con ribavirina, además se realizaron diferentes combinaciones de diferentes cepas, tratadas y no tratadas.
Para ello, se fortificó un vino base de la variedad Palomino Fino con 12,38% v/v hasta 15,5% v/v. Se añadió un volumen de 150 ml en varios frascos de cultivos celulares de acuerdo a las diferentes combinaciones y los controles. Para inocular cada uno de los frascos, se llevó a cabo un conteo de las levaduras crecidas en YPD líquido durante 48 horas mediante el uso de la cámara de Neubauer. Se hizo el cálculo para poder inocular los l necesarios para tener una concentración de las levaduras óptima a 1x106. En el caso de las muestras muy concentradas se hicieron diluciones 10-1 y 10-2 para poder hacer el conteo. Para la mezcla 1:1 se contempló la mitad del volumen de las diferentes cepas o especies de levaduras a inocular. Estos ensayos se realizaron por triplicado.
Los triplicados de cada grupo de los vinos inoculados, se incubaron a temperatura ambiente. La formación de biopelículas de velo de Flor se observó diariamente hasta cubrir la superficie (aproximadamente durante 20 días), de manera que se pudieran obtener datos como el tiempo de su aparición y su consistencia, así como la morfología. Pasados los 20 días, se procedió al aislamiento de levaduras de nuevo, siguiendo el procedimiento del apartado 2.1.2 y se aplicaron las técnicas moleculares de PCR-ITS y RFLP y SSR-PCR, mencionadas en los apartados anteriores, para determinar el porcentaje de patrones de un tipo u otro, y de este modo conocer el grado de implantación de cada cepa enfrentada en cada uno de los ensayos.
2.8 Análisis estadístico
La evaluación estadística de llevó a cabo por medio de la prueba ANOVA (Análisis de varianza), determinándose que existen diferencias estadísticamente significativas entre los valores promedio de los halos de inhibición con un alfa de 0.05.
3 Resultados
3.1 Especies y Cepas de levaduras identificadas
Se aislaron un total de 270 levaduras procedentes de las 9 botas muestreadas en este estudio en la bodega. Mediante la aplicación de la PCR-ITS y RFLP con las enzimas de restricción indicadas en el apartado de Materiales y Métodos, se identificaron dos especies de levaduras; Saccharomyces cerevisiae 90% y Wickerhamomyces anomalus (10%). Tras el análisis por PCR de los microsatélites de las identificadas como S. cerevisiae, se detectaron 3 genotipos diferentes denotados como FV-A1, FV-A2, FV-A4.
Adicionalmente, a estas 270 levaduras se les sumó otras 20 cepas de las especies S. cerevisiae, W. anomalus y Pichia membranifaciens, que fueron aisladas en vinos Finos y Amontillados y caracterizadas previamente en estudios anteriores [7,9]. La nomenclatura de las especies se recoge en la Tabla 2.
Nomenclatura de las cepas utilizadas en el estudio.
3.2 Aislados con Factor Killer en YPD-MB
Los diferentes aislados se sometieron a un screening en medio específico con Azul de Metileno para determinar semi-cualitativamente la capacidad de producir proteínas con factor killer sobre la cepa sensible. Entre los aislados que mostraron ser killer positivos en este estudio solo la cepa FV-A4 de S. cerevisiae mostró esta característica. Respecto a las cepas adicionales de estudios previos, los aislados que mostraron halo de inhibición fueron; las cepas de S. cerevisiae F y G y la especie W. anomalus NsB, así como los aislados de vinos Amontillados de la especie S. cerevisiae AM6 y AM10 y de la especie W. anomalus AM3, AM5, AM7 y AM8. También mostraron factor killer positivo las cepas FI y la cepa FV-A4 aisladas en vinos Finos (Fig. 1). El resto de levaduras ensayadas dieron como resultado ser neutras. Entre las cepas neutras, se seleccionó la cepa A (al ser la mayoritaria en las bodegas del marco de Jerez) como control en las PCR multiplexadas.
Además, se midió en milímetros el diámetro del halo producido por cada una de las cepas que mostraron factor killer positivo. Estos datos numéricos no mostraron diferencias significativas tras aplicar el ANOVA (Tukey, p<0,05)
 |
Figura 1 Ejemplo de placa de Petri con medio YPD-MB y levaduras con factor killer positivo y negativo de acuerdo al halo formado sobre la cepa sensible K0 sembrada en césped. |
3.3 PCR-Multiplex Totivirus en cepas con factor killer
Tras caracterizar el fenotipo killer de las levaduras en estudio de forma cualitativa, se procedió al análisis molecular mediante PCR multiplexada, con el fin de confirmar la presencia de Totivirus involucrados con el fenotipo killer, y que micovirus en concreto eran.
Los resultados mostraron amplificación, correspondientes con unos tamaños de banda de 494 bp y 350 bp, lo cual se puede confirmar la presencia de los Totivirus L-A-lus y M-lus en todas las cepas de velo de Flor que mostraron fenotipo killer en el medio YPD-MB (Fig. 2).
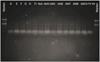 |
Figura 2 Perfil genético de los micovirus presentes en el genoma de las levaduras de velo de Flor analizadas e infectas por Totivirus. Marker = 100 pb. |
3.4 Tratamiento con ribavirina y efectos sobre el fenotipo killer
Todas las levaduras fueron sometidas a diferentes concentraciones de ribavirina (100 μM, 200 μM y 300 μM), incluidas las cepas control, K1 y K0. Tras esto se caracterizó el fenotipo killer en placas Petri con azul de metileno. Los resultados del fenotipo killer en las diferentes cepas tratadas vienen reflejados en la Tabla 3.
El tratamiento a una concentración de 100 μM de ribavirina no resultó ser efectivo en ninguno de los casos, sin embargo, al doblar la concentración (200 μM), fue efectivo en todas las cepas de levaduras a excepción de la cepa FV-A4 aislada en vinos Finos, las cepas aisladas en vinos Amontillados,y la cepa NsB de vinos Finos. Aplicando una concentración aún mayor de ribavirina (300 μM), todas las cepas se volvieron sensibles a excepción de las levaduras no-Saccharomyces que mostraron halos de inhibición aún mayores no tratándolas como se pueden comparar las figuras 1 y 3 y con diferencias significativas tras el ANOVA de los datos obtenidos tras la medición en mm de los halos (Fig. 3).
Tras esta prueba, se procedió a realizar una PCR multiplexada de todas las cepas tratadas con ribavirina a las concentraciones de 200 μM y a 300 μM con el fin de determinar la presencia, o no presencia de los Totivirus de forma cuantitativa.
Tras observar los resultados de la PCR multiplexada del cDNA de las levaduras una vez tratadas con ribavirina, se observa que no aparecen bandas correspondientes a Totivirus en ninguna de las cepas, a excepción de la cepa de W. anomalus en la que sí aparece una banda en torno a 494 pb y otra a 350 bp, confirmando la presencia de los Totivirus L-A-lus y M-lus.
Tratamiento con ribavirina a cada cepa de levadura que mostró factor killer positivo, donde n = neutro y + = positivo.
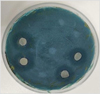 |
Figura 3 Factor killer en placas de YPD-MB de levaduras tratadas con ribavirina a una concentración de 300 μM. |
 |
Figura 4 PCR multiplexada de las diferentes cepas tras el tratamiento con ribavirina a una concentración de 300 μM. Marker = 100 pb. |
3.5 Ensayos de competencia a escala de laboratorio
Respecto a los ensayos de formación de velos de Flor, se pudo observar cómo el velo de Flor se desarrollaba en cada vino diariamente, Las levaduras de velo de Flor que no fueron tratadas, comenzaron a formar el velo antes que aquellas tratadas. Se asume, pues, que aquellas levaduras infectadas por micovirus obtienen ventaja sobre aquellas que no son infectadas por estos tanto en la velocidad de formación del velo como en el grosor y la calidad del mismo (datos no mostrados). Cabe también destacar que en los estudios de competición (1:1) las cepas que mostraron mayor halo en las placas de YPD-MB se impusieron a las que fueron neutras en cuanto ocupando entre el 70-90% del velo de Flor frente a las cepas neutras.
4 Discusión y Conclusiones
A diferencia de muchos virus, los virus que infectan a levaduras Saccharomyces son exclusivamente intracelulares, y no se transmiten de forma extracelular entre sus huéspedes. Para transmitirse de manera exitosa a una población no infectada, los virus, retrotransposones y plásmidos deben iniciar una infección persistente, y permitir a las levaduras Saccharomyces reproducirse de manera sexual mediante fusión célula-célula [15,16]. Esta persistencia a largo plazo ha permitido, con toda probabilidad, coevolucionar con sus huéspedes durante largos periodos de tiempo [17–19]. Podemos encontrar cuatro tipos de elementos de ácido nucleico, que pueden infectar de forma simultánea a levaduras Saccharomyces, siendo estos Narnavirus, retrotransposones Ty, y plásmidos de 2 μm y los Totivirus, siendo estos últimos los de interés para este estudio.
Las cepas infectadas por estos micovirus permanecerán crónicamente infectadas, dando lugar a cepas secretadoras de toxinas, así como al conocido como fenotipo killer, el cual está asociado a la presencia en el citoplasma de las levaduras de los dsRNA de los correspondientes virus. En Saccharomyces cerevisae, este fenotipo killer estará provocado, en concreto, por virus conocidos como L-A y M, de la familia Totiviridae [20]. Se han identificado Totivirus putativos en levaduras del género Saccharomyces por detección de sus dsRNAs, habiéndose encontrado también en S. cerevisiae, siendo estos virus tales como ScV-L-A, ScV-L-A28, ScV-L-A-lus y ScV-L-BC [14,21].
Bajo condiciones normales, las levaduras infectadas por los virus L-A no manifiestan síntomas de infección, pero este virus suele estar acompañado por la presencia del virus satélite M, que codifica para la toxina K, la cual posee capacidad de matar levaduras sensibles a la misma, concediéndole a la levadura un fenotipo killer fácilmente identificable. Estos virus satélites de dsRNA son ácidos nucleicos no autónomos que se replican en el citosol de las levaduras. Para persistir en levaduras Saccharomyces empaquetan y replican su material genético (de unas 500-3000 pb) mediante el secuestro de las proteínas codificadas por los Totivirus. Algunos virus satélites no codifican ninguna proteína, si no solo las secuencias de RNA que posibilitan su encapsidación y replicación. Sin embargo, otros virus satélites pueden codificar las toxinas killer, que serán traducidas, procesadas post-traducción, y excretadas por las levaduras. Las proteínas killer pre-procesadas serán necesarias para la inmunidad de la levadura a las toxinas del mismo tipo. La producción de toxina K por parte de cepas de S. cerevisiae es un carácter que favorece la supervivencia la levadura huésped, pudiendo ser fácilmente caracterizada analizando los halos de inhibición que producen las colonias de las levaduras infectadas sobre placas sembradas con levaduras sensibles. Hay varias cepas con actividad killer descritas, incluyendo K1, K2, Klus y K28 [22]. Cada una de estas cepas depende de su respectivo virus satélite M, y su asociado L-A helper, L-A 1/M1, L-A2/M2, L-A28/M28 y L-Alus/Mlus [23]. Sin embargo, por primera vez, en este estudio, se describe la presencia de estos tipos de Totivirus asociados a las levaduras de velo de Flor.
Las toxinas killer mejor caracterizadas respecto a sus determinantes genéticos, características bioquímicas, objetivos moleculares en las células sensibles y mecanismos de acción son K1, K2, y K28 de S. cerevisiae, la zymocina de Kluyveromyces lactis, PMKT y PKMT2 de Pichia membranifaciens, PaKT de Wyckerhamomyces anomalus, HM-1 de Cyberlindera mrakii, y Kpkt de Tetrapisispora phaffii [14,24-25]. Para el resto de toxinas, en general la localización de los determinantes genéticos de las toxinas killer han sido ya descritos, si bien aún se necesita saber mucho más con respecto a sus modos de acción, objetivos moleculares dentro de células sensibles e inmunidad a la toxina y especialmente en su influencia en la formación del velo de Flor en vinos fortificados con bajo pH y altas concentraciones de etanol.
La mayoría de toxinas killer son estables a pH ácido, si bien algunas mantienen su actividad a un amplio rango de pH, como HM-1. [26]. Respecto al rango de temperatura de su actividad, este también varía en función al hábitat de la levadura killer en cuestión. Por ejemplo, las levaduras marinas producen una toxina killer con una temperatura óptima de 15 ºC, mientras que aquellas producidas por cepas de suelo, como T. Phaffi. generalmente, la actividad de las toxinas killer se inhibe a temperaturas superiores a 40 ºC [14]. Además, algunas toxinas muestran actividades superiores en presencia de NaCl, siendo esto especialmente importante en fermentaciones de aceitunas y en levaduras típicas de ambientes marinos [27–28].
En nuestro estudio, empleamos además ribavirina para tratar a las levaduras objeto de estudio, sin embargo, ésta no fue efectiva en el caso de las levaduras de especies no-Saccharomyces cuando se realizaron pruebas en placas de YPD-MB, lo cual este antiviral podría estar influyendo de forma positiva en el aumento de actividad killer de estas levaduras. Se observó, en el caso de la cepa NsB, una mayor presencia de Totivirus tras la amplificación por PCR de los mismos, no ocurrió así en el resto de cepas no-Saccharomyces. Serán necesarios más estudios para comprender este fenómeno. Cabe destacar que la mayoría de las levaduras de velo de Flor de género Saccharomyces empleadas en este estudio presentaron carácter neutro en el screening inicial, a excepción de los genotipos F y G, que curiosamente fueron exclusivos en dos bodegas según argumentaron Ruiz-Muñoz et al. [7]. El empleo del antiviral rivabirina parece ser tóxico a concentraciones mayores a 100 µM, privando a las mismas de los Totivirus (tal y como muestran los resultados obtenidos por PCR multiplexada) y volviendo a estas levaduras sensibles a las proteínas killer, y en consecuencia incapaces de imponerse durante la formación del velo de Flor. Esto nos permitiría controlar los procesos de formación de velo de Flor a escala industrial y hacer una selección de las levaduras deseadas o eliminar las indeseadas, no obstante, serían necesarios estudios más profundos.
Los resultados obtenidos en este estudio preliminar parecen indicar que la presencia de micovirus en las levaduras pueden cursar de forma asintomáticas en las mismas (factor killer neutro) confiriéndoles de resistencia a otras toxinas killer o dotándolas de carácter killer positivo que a su vez les permite ser dominantes en el complejo de un velo de Flor. Además, la presencia de estos Totivirus, podrían estar relacionados con la supervivencia y adaptación de las levaduras de velo de Flor a las condiciones de estrés del medio en el que se forman, vinos con un pH bajo y una alta concentración de etanol.
References
- R.B. Wickner, T. Fujimura, R. Esteban, Adv Virus Res 86, 1–36 (2013) [CrossRef] [PubMed] [Google Scholar]
- J.I. Maruyama, K. Kitamoto, Microorganisms 7(12), 639 (2019) [CrossRef] [PubMed] [Google Scholar]
- M.W. Taylor, P.S. Tsai, Proc Natl Acad Sci U S A 86(23), 9275–9279 (1989) [Google Scholar]
- C. Garmendia-Torres, A.L. Goldstein, Y.E. Fritz, et al., PLoS Pathog 17(3), e1009476 (2021) [CrossRef] [PubMed] [Google Scholar]
- K. Seppälä, P. Lehtovaara, E. Vartiainen, et al., FEMS Yeast Res 12(7), 747–757 (2012) [Google Scholar]
- S.A. Ghabrial, J.R. Castón, D. Jiang, et al. “Totiviridae”. En: Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses. Elsevier (2011), p. 543-549 [Google Scholar]
- M. Ruiz-Muñoz, G. Cordero-Bueso, F. Benítez-Trujillo, S. Martínez, F. Pérez, J.M. Cantoral, Food Microbiol. 92, 103553 (2020) [CrossRef] [Google Scholar]
- M. Ruiz-Muñoz, M. Hernández-Fernández, G. Cordero-Bueso, S. Martínez-Verdugo, F. Pérez, J.M. Cantoral, Fermentation 8, 456 (2022) [CrossRef] [Google Scholar]
- M.E. Rodríguez, J.J. Infante, J.J. Mesa, L. Rebordinos, J.M. Cantoral, Open Biotechnol J. 7, 23–29 (2013) [CrossRef] [Google Scholar]
- B. Esteve-Zarzoso, M. T. Fernández-Espinar, A. Querol, Antonie van Leeuwenhoek 85(2), 151–158, (2004) [CrossRef] [PubMed] [Google Scholar]
- J. Sabate, J. Cano, B. Esteve-Zarzoso, J. M. Guillamón, Microbiol Res, 157(4), 267–274 (2002) [CrossRef] [Google Scholar]
- Vaudano y E. Garcia-Moruno, Food Microbiol 25(1), 56–64 (2008) [CrossRef] [PubMed] [Google Scholar]
- Vaudano, G. Quinterno, A. Costantini, L. Pulcini, E. Pessione, y E. Garcia-Moruno, Int J Food Microbiol 289, 154–161 (2019) [CrossRef] [PubMed] [Google Scholar]
- J. Quintero-Blanco, J. Jimenez, A. Garzón. Appl Environ Microbiol 88(4), e02213-21 (2022) [CrossRef] [PubMed] [Google Scholar]
- C. Zeyl, G. Bell, D.M. Green. Genetics 143(4), 1567–1577 (1996) [CrossRef] [PubMed] [Google Scholar]
- A.B. Futcher, E. Reid, D.A. Hickey. Genetics 118(3), 411–415 (1988) [CrossRef] [PubMed] [Google Scholar]
- G.I. Naumov, Y.V. Ivannikova, I.Y. Chernov, E.S. Naumova. Microbiology 78(2), 208–213 (2009) [CrossRef] [Google Scholar]
- P.K. Strope, D.A. Skelly, S.G. Kozmin, G. Mahadevan, E.A. Stone, P.M. Magwene, F.S. Dietrich, J.H. McCusker. Genome Res. 25, 762–774 (2015) [CrossRef] [PubMed] [Google Scholar]
- G. Liti, A. Peruffo, S.A. James, I.N. Roberts, E.J. Louis. Yeast 22(3), 177–192 (2005) [CrossRef] [PubMed] [Google Scholar]
- E.A. Berry, E.A. Bevan. “A new species of double-stranded RNA from yeast." Nature 239, 279-280 (1972) [CrossRef] [PubMed] [Google Scholar]
- C.-M. Park, J.D. Lopinski, J. Masuda, T.-H. Tzeng, J.A. Bruenn. Virology 216(2), 451–454 (1996) [CrossRef] [Google Scholar]
- M. Ramírez, R. Velázquez, A. López-Piñeiro, B. Naranjo, F. Roig, C. Llorens. Toxins 9, 292 (2017) [CrossRef] [PubMed] [Google Scholar]
- N. Rodríguez-Cousiño, P. Gómez, R. Esteban. Toxins 9, 313 (2017) [CrossRef] [PubMed] [Google Scholar]
- A. Santos, D. Marquina. Yeast 21, 151–162 (2004) [CrossRef] [PubMed] [Google Scholar]
- S. Kashara, S.B. Inoue, I. Mio et al. FEBS Lett. 348, 27–32 (1994) [CrossRef] [PubMed] [Google Scholar]
- T. Yamamoto, T. Hiratani, H. Hirata et al. FEBS Lett. 197, 50–54 (1986) [CrossRef] [PubMed] [Google Scholar]
- P. Llorente, D. Marquina, A. Santos, et al. Appl Environ Microbiol. 63, 1165–1167 (1997) [CrossRef] [PubMed] [Google Scholar]
- G.L. Liu, K. Wang, M.X. Hua, et al. Proc Biochem. 47, 822–827 (2012) [CrossRef] [Google Scholar]
All Tables
Relación de primers utilizados para la amplificación del genoma vírico relativo a los Totivirus de levaduras.
Tratamiento con ribavirina a cada cepa de levadura que mostró factor killer positivo, donde n = neutro y + = positivo.
All Figures
 |
Figura 1 Ejemplo de placa de Petri con medio YPD-MB y levaduras con factor killer positivo y negativo de acuerdo al halo formado sobre la cepa sensible K0 sembrada en césped. |
| In the text | |
 |
Figura 2 Perfil genético de los micovirus presentes en el genoma de las levaduras de velo de Flor analizadas e infectas por Totivirus. Marker = 100 pb. |
| In the text | |
 |
Figura 3 Factor killer en placas de YPD-MB de levaduras tratadas con ribavirina a una concentración de 300 μM. |
| In the text | |
 |
Figura 4 PCR multiplexada de las diferentes cepas tras el tratamiento con ribavirina a una concentración de 300 μM. Marker = 100 pb. |
| In the text | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.



